quimico
2011-12-19 17:33
1. Introduzione
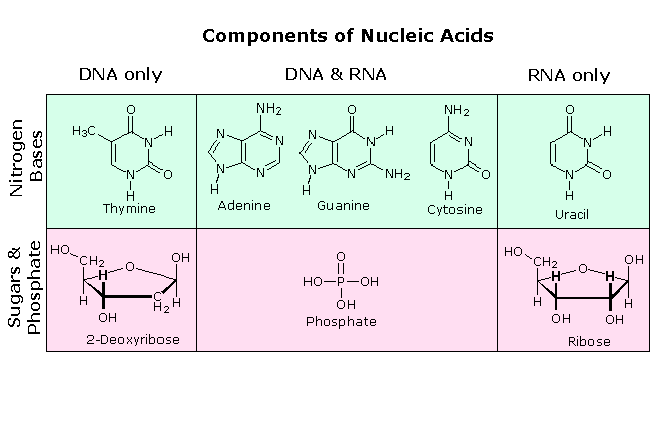
Il primo isolamento di quello che ora noi sappiamo essere il DNA fu portato a termine da Johann Friedrich Miescher circa nel 1870. Riportò la scoperta di una sostanza debolmente acida di funzione ignota nel nucleo dei globuli bianchi nel sangue, e chiamò questo materiale "nucleina". Pochi anni più tardi, Miescher separò la nucleina nei componenti proteina ed acido nucleico. Negli anni '20 gli acidi nucleici vennero scoperti essere i componenti maggioritari dei cromosomi, piccoli corpi recanti geni, nei nuclei di cellule complesse. L'analisi elementale degli acidi nucleici mostrò la presenza di fosforo, oltre ai soliti C, H, N & O. Diversamente dalle proteine, gli acidi nucleici non contengono zolfo. L'idrolisi completa degli acidi nucleici cromosomiali dava fosfato inorganico, 2-deossiribosio (un zucchero fino ad allora ignoto) e quattro differenti basi eterocicliche (mostrate nello schema sotto). Per evidenziale l'insolita componente 2-deossiribosio, gli acidi nucleici cromosomiali sono chiamati acidi deossiribonucleici, abbreviato DNA. Gli analoghi acidi nucleici in cui la componente zucchero è il ribosio sono chiamati acidi ribonucleici, abbreviato RNA. Il carattere acido degli acidi nucleici venne attribuito alla metà acido fosforico.
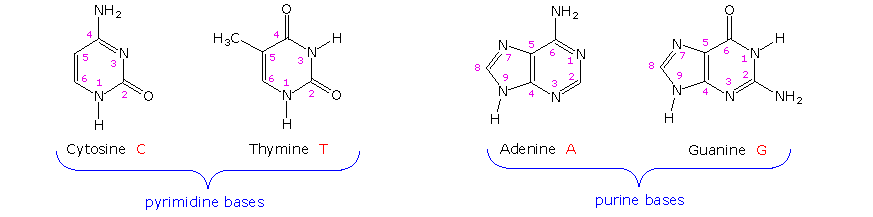
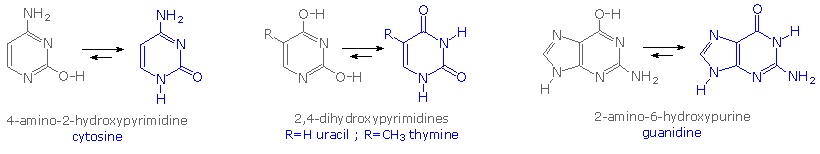
Le due basi monocicliche mostrate qui sono classificate come pirimidine, e le due basi bicicliche sono purine. Ognuna ha almeno un sito N-H a cui può essere attaccato un sostituente organico. Esse sono tutte basi polifunzionali, e possono esistere in forme tautomeriche.
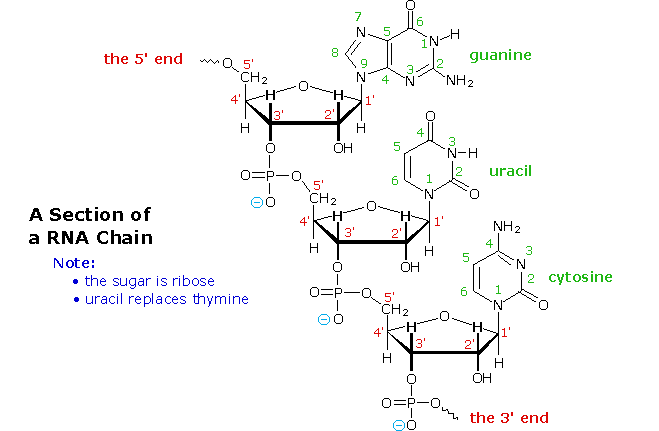
L'idrolisi base catalizzata del DNA ha portato quali prodotti a quattro nucleosidi, che si sono dimostrati essere N-glicosidi del 2'-deossiribosio combinati con le amine eterocicliche. Le strutture ed i nomi di questi nucleosidi sono mostrati sotto.
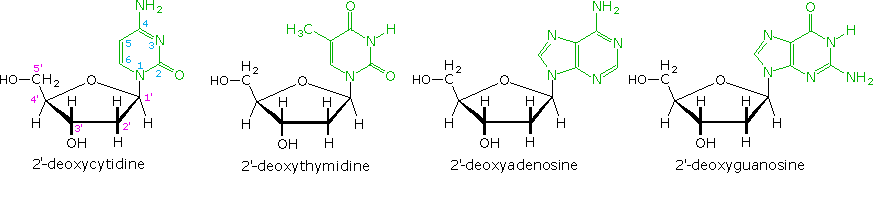
Le basi che compongono tali nucleosidi sono colorate in verde, e lo zucchero è nero. Come notato nella struttura della 2'-deossicitidina sulla sinistra, la numerazione dei carboni dello zucchero fa uso dei numeri adatti per distinguerli dai siti delle base eterociclica. I corrispondenti N-glicosidi del comune zucchero ribosio sono i building blocks dell'RNA, e sono chiamati adenosina, citidina, guanosina ed uridina (un analogo della timidina cui manca il gruppo metile).
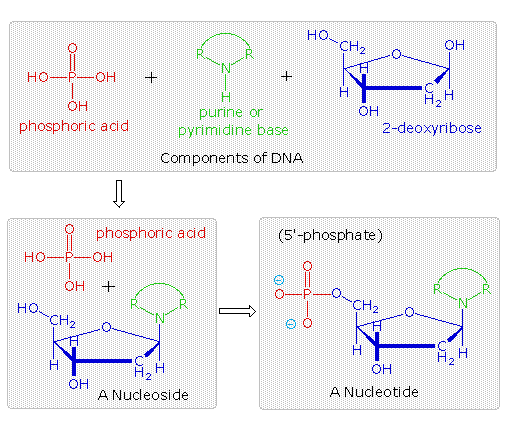
Da questa prova, gli acidi nucleici possono essere formulati come copolimeri che si alternano di acido fosforico (P) e nucleosidi (N), come mostrato:
~ P – N – P – N'– P – N''– P – N'''– P – N ~
All'inizio i quattro nucleosidi, distinti dagli apici in questra formula grezza, vennero assunti essere presenti in uguali quantità, come si risultava dalla struttura uniforme, come quella dell'amido. Comunque, un composto di questo tipo, presumibilmente comune a tutti gli organismi, fu considerato troppo semplice per possedere l'informazione ereditaria che risiede nei cromosomi. Questo punto di vista fu messo alla prova nel 1944, quando Oswald Avery e colleghi dimostrarono che il DNA batterico era probabilmente l'agente genetico che portava l'informazione da un organismo all'altro in un processo chiamato "trasformazione". Egli concluse che "gli acidi nucleici devono essere considerati possedere specificità biologica, la cui base chimica è finora indeterminata." Nonostante questa scoperta, molti scienziati continuarono a credere che le proteine cromosomiali, che sono diverse tra le specie, tra gli individui, ed anche all'interno di dati organismi, erano il luogo dell'informazione genetica di un organismo.
Dovrebbe essere notato che gli organismi unicellulari come i batteri non hanno un nucleo ben definito. Invece, il loro unico cromosoma è associato a proteine specifiche in una regione chiamata "nucleoide". Nonostante ciò, il DNA dai batteri ha la stessa composizione e la stessa struttura generale come quella degli organismi pluricellulari, inclusi gli esseri umani.
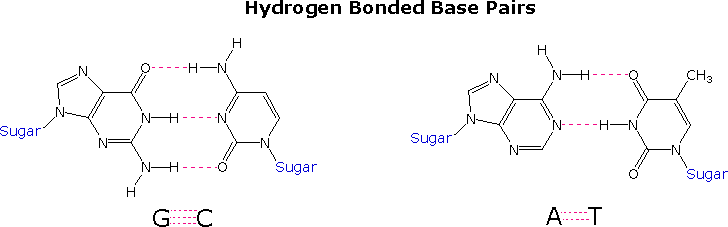
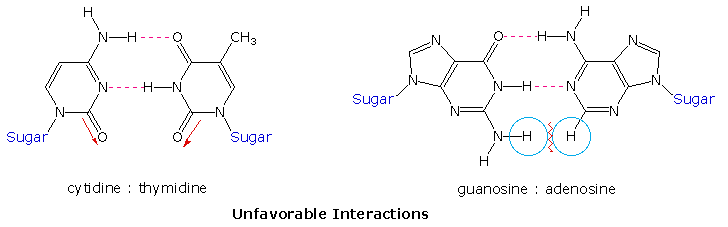
I punti di vista circa il ruolo del DNA nell'eridarietà cambiarono verso la fine degli anni '40 e gli inizi degli anni '50. Conducendo un'attenta analisi del DNA da molte fonti, Erwin Chargaff trovò che la sua composizione era specifica per una data specie. Inoltre, scoprì che la quantità di adenina (A) uguagliava sempre la quantità di timina (T), e che la quantità di guanina (G) uguagliava sempre la quantità di citosina (C), a prescindere dalla fonte del DNA. Il rapporto (A+T)/(C+G) variava da 2.70 a 0.35. Gli ultimi due organismi sono batteri.
Distribuzione delle basi nucleosidiche nel DNA
Organismo - Composizione delle basi A G T C (mole %) - Rapporti tra le basi A/T G/C - Rapporto (A+T)/(G+C)
Umano 30.9 19.9 29.4 19.8 1.05 1.00 1.52
Pollo 28.8 20.5 29.2 21.5 1.02 0.95 1.38
Lievito 31.3 18.7 32.9 17.1 0.95 1.09 1.79
Clostridium perfringens 36.9 14.0 36.3 12.8 1.01 1.09 2.70
Sarcina lutea 13.4 37.1 12.4 37.1 1.08 1.00 0.35
In un secondo studio critico, Alfred Hershey e Martha Chase mostrarono che quando un batterio viene infettato e geneticamente trasformato da un virus, almeno l'80% del DNA virale entra nella cellula batterica ed almeno l'80% della proteina virale rimane fuori. Assieme alle scoperte di Chargaff questo lavoro stabilì che il DNA è il depositario delle caratteristiche uniche, genetiche di un organismo.
I seguenti utenti ringraziano quimico per questo messaggio: ClaudioG.
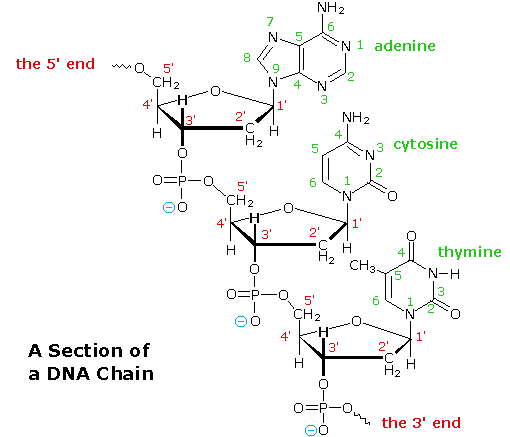
 è diversa dall'altra (3'
è diversa dall'altra (3'